Smart-seq
Smart-seq技术是由美国和瑞典科学家共同开发的一种单细胞转录组测序技术,其能够检测mRNA的全长。
测序原理
用莫洛尼鼠类白血病病毒逆转录酶(moloney murine leukaemia virus reverse transcriptase,MMLVRT)逆转录全部RNA,然后加入PCR引物,进行常规PCR扩增,再进一步将扩增的cDNA打断,用于构建标准的Illumina测序文库。
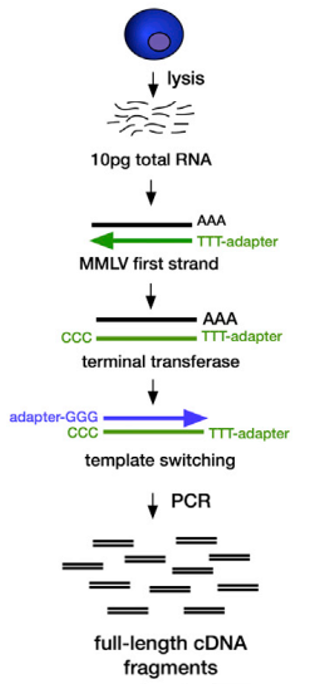
Smart-seq关键技术
- CDS引物:(5’AAGCAGTGGTATCAACGCAGAGTACT(30)VN3’,V代表A、C或G,N代表A、T、C或G):两个末端核苷酸将引物锚定在poly(A)尾巴的开始处,这样就保证了逆转录的起始位置正好是mRNA的3’端的序列终止位置。
- 引物2:由一段通用序列及它的3’端是3个非脱氧的G碱基构成(RNA的G碱基),这个引物可以与新合成的cDNA的3’端的几个C碱基互补杂交。
- MMLVRT:同时具有模板转换和末端转移酶的活性,其在转录到mRNA的5’端末端的时候,会在新合成的cDNA的3’末端,多出几个C碱基。
Smart-seq测序流程
- 分离获得单个细胞,并将单个细胞转移至低渗裂解缓冲溶液(裂解液中含有RNase抑制剂)中进行裂解,转录组释放于裂解液中。
- 加入MMLV逆转录酶、dNTP、oligo(dT)VN Primer、MgCl2等,对mRNAs进行反转录,获得cDNA第一条链,同时由于MMLVRT的末端转移酶活性会向cDNA的5’末端加上非模板化的胞嘧啶(C)残基。
- 引物2对所加的C碱基进行互补配对,然后引导MMLVRT再次发挥聚合作用,得到双链cDNA。
- cDNA合成之后,进行12-18个循环的cDNA PCR扩增(100 pg的总RNA,需要15个循环,对于10 pg的总RNA,可以进行18个循环),将扩增的cDNA用于构建标准的Illumina测序文库。
优点
- 完整阅读mRNA5’端。
- 可用于筛选SNPs。
- 不需要知道mRNA的序列。
- 使用低于50pg的起始材料。
- 高水平的可定位序列。
- 突出优点是可以筛选单核苷酸多态性(SNPs)和变异体。
缺点
- 不稳定,低水平表达的转录组可能丢失。
- 转录本长度偏向性,对超过4Kb的序列不能高效转录。
- 并非链特异的。
- 价格较高。
Smart-seq2
Smart-seq2是经Smart-seq改进的一种测序方法,也目前最常用的单细胞转录组技术之一。
测序原理
通过设计oligo(dT)VN Primer作为逆转录引物,利用MMLVRT的模板转换活性,在cDNA的3’端添加一段接头序列,通过该接头序列进行反转录,生成cDNA第一条链。当逆转录酶到达mRNA5’末端时,会连续在末端添加几个胞嘧啶(C)残基。然后添加TSO引物,退火后结合在第一条链的3’端与poly(C)突出杂交,合成第二条链。这样得到的cDNA经过PCR扩增,获得纳克级的DNA,纯化后可用于测序。
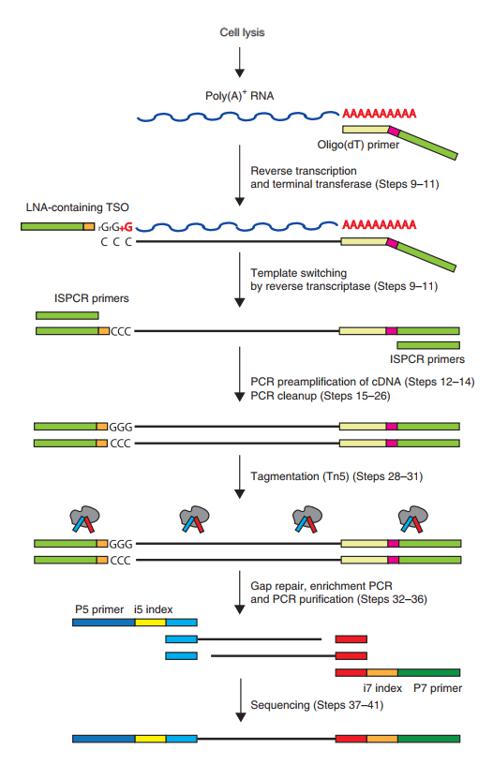
Smart-seq2关键技术
甜菜碱(N,N,N三甲基甘氨酸)是一种高效的甲基供体。在反转录体系中加入甜菜碱,其能够增加转录所需酶(蛋白质)的热稳定性,并通过破坏DNA螺旋的稳定性降低碱基对DNA热融化转变的依赖性。此外,甜菜碱还可以使cDNA的形成更加完整。一些RNA有着例如发夹结构、环形结构等二级结构,影响逆转录酶与其结合,加入甜菜碱可解决这一问题,最终得到完整的cDNA文库。此外,在加入甜菜碱的同时增加Mg2+的浓度也可在一定程度上提高cDNA产量。
TSO引物序列(5’-AAGCAGTGGTATCAACGCAGAGTACATrGrG+G-3’):在5’端带有1个通用引物序列,而在3’末端,有两个核糖鸟苷(rG)和一个锁核苷酸(Locked Nucleic Acid,LNA)修饰的鸟苷(G),可以促进模板转换,进而扩增出完整的cDNA序列。
Smart-seq2测序流程
- 分离出单细胞,并用裂解液裂解细胞。
- 在裂解液中加入MMLV逆转录酶、游离的dNTPs、oligo(dT)VN Primer、甜菜碱、MgCl2等,通过oligo(dT)VN Primer启动mRNAs反转录,获得cDNA第一条链。
- RT反应通常在42°C进行90分钟,但是某些RNA形成二级结构(例如发夹或环),由于空间位阻,可能导致酶终止链延长。通过添加海藻糖和甜菜碱,可以在某种程度上克服这种不良影响。
- 当反转录到达RNA5’末端后,MMLV逆转录酶的末端转换活性会在cDNA末端加非模板编码的核苷酸,通常是dCTP。
- 加入模板转换引物TSO,当反转录到达mRNA的5’末端,TSO3’的r(G)3和cDNA序列中富含dC的序列依靠互补作用促进模板转换。MMLV反转录酶会转录这个寡核苷酸序列,进行后续PCR扩增,获得双链cDNA。
- cDNA合成之后,加入常规PCR引物,进行常规PCR扩增,将扩增的cDNA打断,构建标准的Illumina测序文库。
优点
能够得到全长cDNA。用于分析可变剪切等。
缺点
只分析带Poly(A)的RNA。
Smart-seq与Smart-seq2的区别
- Smart-seq2与Smart-seq相比,使用了锁核酸(locked nucleic acid,LNA)、更高浓度的MgCl2以及甜菜碱。
- Smart-seq2不需要纯化步骤,比Smart-seq产量更高。
- Smart-seq2只能对具有polt-(A)尾巴的mRNA进行扩增和检测。
参考文献
[1] Picelli S , Faridani O R , Bj?Rklund ? K , et al. Full-length RNA-seq from single cells using Smart-seq2[J]. Nature Protocols, 2014, 9(1):171-181.
[2] Wang Y , Navin N . Advances and Applications of Single-Cell Sequencing Technologies[J]. Molecular Cell, 2015, 58(4):598-609.
